
近日,MindSpore社区与北京大学生物医学前沿创新中心(BIOPIC)、北京大学化学与分子工程学院、深圳湾实验室高毅勤教授课题组联合推出蛋白质多序列比对(Protein MSA)数据集,希望在标准化的数据集基础上,支撑研究人员开发先进的AI模型,加深对蛋白质结构、功能和进化的认知,并进行蛋白设计与改造。此数据集的相关代码及数据集说明已依托于华为全场景AI框架MindSpore进行开源开放、定期扩展与维护,旨在为全世界相关的产、学、研团队提供优质的数据共享解决方案。
01 规模最大、覆盖度最广的开源数据集
本次开源的ProteinMSA数据集完全覆盖最新版本(2021年2月发布)的UniRef50数据库中的蛋白质序列,采用学术界的“金标准”搜索方法,对约0.5亿条蛋白序列进行了充分的MSA搜索与比对(MSA平均深度大于1000),是目前世界范围内规模最大、参考数据集最新、覆盖度最广的开源蛋白质MSA数据集(之前最大的开源MSA数据集包含10万个蛋白MSA)【1】。
人类已知的蛋白质序列已经超过4.4亿条,但仅凭这些蛋白质单序列数据库,很难了解蛋白之间的关系。Protein MSA数据库是一个对不同蛋白质序列之间的关系进行了标记的大规模“关系型”数据库,被标记为关联的蛋白质序列之间的相似度、进化关系、突变所在位点的分布等信息对蛋白质结构和功能的预测极为重要。
为了更好地服务于跨领域的研究人员,Protein MSA数据集将被组织成具有多重形态的数据格式。原始数据集(近30T)将以UniRef系列数据库【2】和UniClust数据库【3】的标准文本形式存储放,并按照序列长度进行分割与压缩。为了便于AI领域的研究人员直接使用,Protein MSA数据集还会将文本格式的数据集转化为浮点数张量类型压缩存储,并对已有的AI框架如MindSpore进行数据接口的支持。
02 业内首创数据License审核流程,助力MSA数据集合规发布
开放数据集提升了数据这一重要资源在AI领域的可用性,但是和开源软件一样,数据集对外开放同样需要遵从原作者的许可,否则将有可能侵犯他人的知识产权。
本次MSA数据集发布引入了由MindSpore数据合规SIG组建立的国内首创的数据License审核流程。由数据集作者与MindSpore开源及法务专家团队共同对MSA数据集的License合规进行审查,最终团队基于MSA数据集与UniRef50,UniClust30等数据集兼容性考虑,确定采用CC BY-SA 4.0 License作为MSA数据集授权许可。具体授权条款可参考:https://creativecommons.org/licenses/by-sa/4.0/deed.zh
03 Protein MSA开源数据集初探
-
Potiein MSA数据库简介
针对蛋白质的多序列比对(multiple sequence alignment; MSA)是研究蛋白质的结构、功能和进化关系等问题的重要方法。MSA数据中蕴含了构成蛋白质的氨基酸序列中的保守性质(conservation)、协同突变(co-evolution)和功能与物种进化关系(phylogenetics)的相关信息(img cr Sergey.O)。
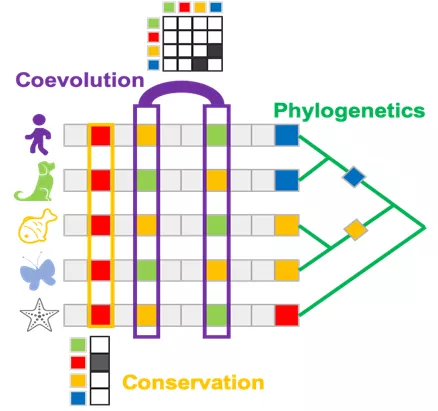
人类已知的存在于自然界中的蛋白质序列数目已经上亿并在快速增长,但仅凭这些蛋白质单序列的数据很难了解蛋白之间的关系。Protein MSA数据库,就是一个对不同蛋白质序列之间的关系进行了标记的大规模“关系型”数据库,被标记为关联的蛋白质序列之间的相似度、进化关系和突变所在位点的分布等信息对蛋白质结构和功能的预测极为重要。例如在AlphaFold2模型中,目标蛋白序列的MSA信息就是预测结构的必要输入信息之一。
-
数据库建立方法
Protein MSA中的目标序列将几乎完全覆盖最新版本(2021.02发布)的UniRef50数据库中的蛋白质序列,而比对序列来自于最新版本的UniClust30数据库。对于每条来自UniRef50数据库中的目标序列,我们采用HHBlits算法在UniClust30数据库进行搜索和比对,并将检索以文本形式存放于Protein MSA数据仓库下Raw_Data目录下。生成命令如下(参考https://github.com/soedinglab/hh-suite):
hhblits -i <input-file> -d <database-basename> -o <result-file> -oa3m <result-alignment> -cpu 4 -n 3 -v 0pu 4 -n 3 -v 0
-
数据集下载方法
准备华为云账号并登录
1、注册华为云中国站账号后,登录华为云控制台(链接:https://auth.huaweicloud.com),如下图所示,进入“我的凭证”
2、如果你已经有华为云账号,可直接访问 (https://console.huaweicloud.com)登录华为云控制台
使用客户端软件下载数据集
1、安装数据上传客户端软件
当前华为云OBS对象存储服务,提供了2款客户端软件:含视窗操作界面的OBS Browser+, 命令行 工具 客户端 obsutil 。请查看两款软件支撑的操作系统场景,自行选择。
2、准备工作,获取账号ak/sk
在“我的凭证”页面,如下图所示,进入访问密钥的页签,可以创建生成ak/sk访问密钥,创建成功后,保存到本地(CSV文件),后续需要用到。
使用OBS Browser+客户端软件进行数据集的下载
1、登录
打开obsBrowser+, 将事先准备好的 ak(即AccessKey ID)、sk(SecretAccess Key)(保存的CSV文件中),按照下图填写,账号名仅用于区分您在本地登录OBSBrowser+的不同账号,与注册的云服务账号无关,也无需一致。
2、挂载外部桶
如下图所示,输入桶名:cnnorth4-modelhub-datasets-obsfs-d1opw 进行挂载。
3、下载数据集
进入到MSA_Uniref50文件夹,并选中需要下载的数据集文件夹,进行下载。
使用obsutil命令行工具进行数据集的下载
1、安装命令行工具
安装详细官方指导,请参考华为云文档资料:
https://support.huaweicloud.com/utiltg-obs/obs_11_0003.html 。
此处我们仅提供Ubuntu18.04的操作系统上安装的流水账步骤:
打开你的的Terminal,cd 到你期望的安装目录,依次执行如下四个命令:
wget https://obs-community.obs.cn-north-1.myhuaweicloud.com/obsutil/current/obsutil_linux_amd64.tar.gztar -xzvf obsutil_linux_amd64.tar.gzcd obsutil_linux_amd64_*chmod 755 obsutil
执行成功后,如下图所示:
2、初始化配置
执行如下命令,其中ak,sk是你在之前步骤中已经准备好的账号鉴权信息。
endpoint固定为:https://obs.cn-north-4.myhuaweicloud.com
./obsutil config -i=ak -k=sk -e=endpoint
执行成功后,如下图所示:

3、列表文件
详细官方指导,请参考华为云文档资料:https://support.huaweicloud.com/utiltg-obs/obs_11_0014.html
此处我们仅提供Ubuntu 18.04的操作系统上的流水账步骤:
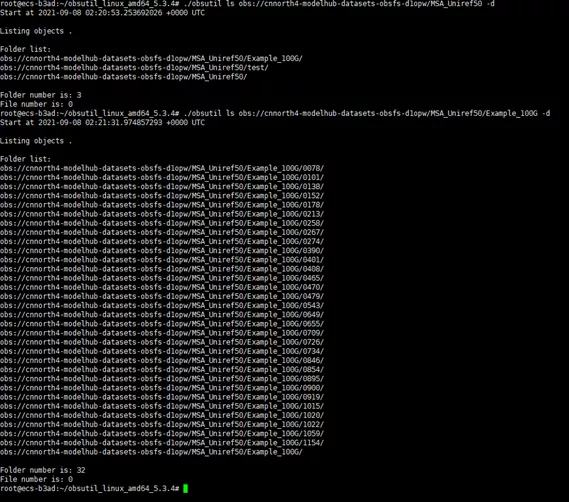
打开你的Terminal,cd 到你obsutil的安装目录,通过以下命令列表数据集内的文件/文件夹,执行效果如下图所示。
./obsutil ls obs://cnnorth4-modelhub-datasets-obsfs-d1opw/MSA_Uniref50 -d
4、递归下载目录
详细官方指导,请参考华为云文档资料:https://support.huaweicloud.com/utiltg-obs/obs_11_0018.html
此处我们仅提供Ubuntu 18.04的操作系统上流水账步骤:
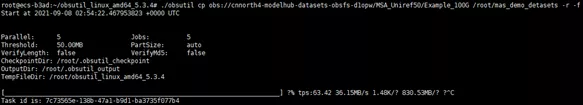
打开你的Terminal,cd 到你obsutil的安装目录,通过以下命令递归下载目录,执行效果如下图所示。
./obsutil cp obs://cnnorth4-modelhub-datasets-obsfs-d1opw/MSA_Uniref50/Example_100G /root/mas_demo_detasets -r -f
5、增量同步递归下载目录
使用场景:
①当上述递归下载目录出现有些文件下载失败的场景,可以使用这个命令,自动检测缺失的文件,并下载
②下载数据的电脑因为意外重启了,需要继续断点续传
详细官方指导,请参考华为云文档资料:https://support.huaweicloud.com/utiltg-obs/obs_11_0043.html
此处我们仅提供Ubuntu 18.04的操作系统上流水账步骤:
打开你的Terminal,cd 到你obsutil的安装目录,通过以下命令增量同步递归下载目录,执行效果如下图所示
./obsutil sync obs://cnnorth4-modelhub-datasets-obsfs-d1opw/MSA_Uniref50/Example_100G /root/mas_demo_detasets
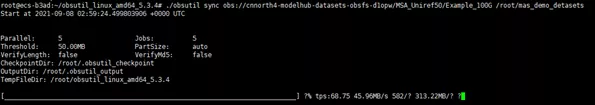
-
维护、更新与社区贡献方式
使用客户端进行数据集上传
按照下载数据集的方法进行客户端的安装获取,进入登录页面后可以直接选择本地数据集进行上传操作注意点:
①对象权限:需要选择 “桶拥有者完全控制”
②存储类别:选择“标准存储”
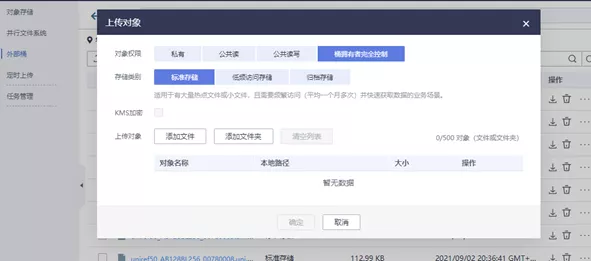
使用obsutil命令行工具进行数据集的上传
安装详情可参考:https://support.huaweicloud.com/utiltg-obs/obs_11_0003.html
下面以 linux 操作系统为例,进行配置:
1、安装
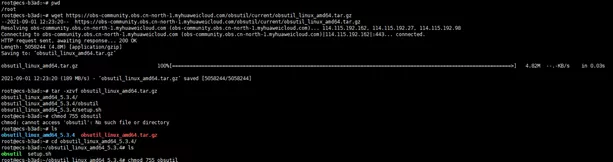
打开命令行工具,cd 到你的安装目录,依次执行如下四个命令:
wget https://obs-community.obs.cn-north-1.myhuaweicloud.com/obsutil/current/obsutil_linux_amd64.tar.gztar -xzvf obsutil_linux_amd64.tar.gzcd obsutil_linux_amd64_*chmod 755 obsutil
执行成功后,如下图所示:
2、初始化配置
执行如下命令,其中ak, sk是你在之前步骤中已经准备好的账号鉴权信息。endpoint固定为:https://obs.cn-north-4.myhuaweicloud.com
./obsutil config -i=ak -k=sk -e=endpoint
执行成功后,如下图所示:

欢迎参与更多贡献,代码,文档不限!
04 展望未来
高毅勤教授表示:“我们鼓励并期待来自生物信息学、数据科学和AI研究等领域的专家和人才充分碰撞与合作,引入、改进或设计全新的AI模型,来充分地挖掘Protein MSA数据集中所隐藏的‘自然的秘密’”。
从科学的角度看,MSA的数量和质量很大程度上影响了目前最先进结构模型的预测速度和精度,而且产生MSA的非参数化算法仍是诸多蛋白预测方法中决定速度的主要步骤之一。因此,Protein MSA数据库本身可以作为这些结构预测模型的预训练材料,用来挖掘序列信息甚至快速生成新的序列特征,这对解决研究、设计蛋白质中所面临的高变异序列和孤儿序列等问题具有巨大的潜在价值。
此次数据库的发布外,MindSpore社区也与北京大学高毅勤课题组联合开发并开源了首个国产分子动力学软件MindSponge,并在8月刚刚举办了国内首场有关开源的人工智能+分子动力学的在线工作坊。

课程回顾 | 公开课MindSpore–SPONGE 暑期线上培训精彩回顾!
未来,MindSpore社区也将牵手更多的学术科研界合作伙伴,在材料、生物、医药等更广泛的科学计算领域打造数据推动的研究新模式。
附:
数据集开源说明:
https://gitee.com/mindspore/mindscience/tree/master/MindSPONGE/protein_msa
数据集下载地址:
https://marketplace.huaweicloud.com/markets/aihub/datasets/detail/?content_id=5802def2-5fbd-40da-85d8-a4541d1c6f1e
【1】AlQuraishi, Mohammed. "ProteinNet: a standardizeddata set for machine learning of protein structure." BMC bioinformatics20.1 (2019): 1-10.
【2】Suzek, B. E., Wang, Y., Huang, H.,McGarvey, P. B., Wu, C. H., & UniProt Consortium. (2015). UniRef clusters:a comprehensive and scalable alternative for improving sequence similaritysearches. Bioinformatics, 31(6), 926-932.
【3】Mirdita M.*, von den Driesch L.*, GaliezC., Martin M. J., Söding J.#, and Steinegger M.#, Uniclust databases ofclustered and deeply annotated protein sequences and alignments, Nucleic AcidsRes. 2016.
